O que é GLM em R?
Modelos lineares generalizados é um subconjunto de modelos de regressão linear e suporta efetivamente distribuições não normais. Para suportar isso, é recomendável usar a função glm (). O GLM funciona bem com uma variável quando a variação não é constante e distribuída normalmente. Uma função de link é definida para transformar a variável de resposta para ajustar-se ao modelo apropriado. Um modelo linear é feito com a família e a fórmula. O modelo GLM possui três componentes principais chamados aleatórios (probabilidade), sistemáticos (preditores lineares) e componentes de link (para a função logit). A vantagem de usar o glm é que eles têm flexibilidade do modelo, não há necessidade de variação constante e esse modelo se ajusta à estimativa de probabilidade máxima e suas proporções. Neste tópico, vamos aprender sobre o GLM em R.
Função GLM
Sintaxe: glm (fórmula, família, dados, pesos, subconjunto, Iniciar = nulo, modelo = TRUE, método = ””…)
Aqui, os tipos de família (incluem tipos de modelo) incluem binomial, Poisson, Gaussiano, gama, quase. Cada distribuição executa um uso diferente e pode ser usada na classificação e na previsão. E quando o modelo é gaussiano, a resposta deve ser um número inteiro real.
E quando o modelo é binomial, a resposta deve ser classes com valores binários.
E quando o modelo é Poisson, a resposta deve ser não negativa com um valor numérico.
E quando o modelo é gama, a resposta deve ser um valor numérico positivo.
glm.fit () - Para ajustar um modelo
Lrfit () - indica ajuste de regressão logística.
update () - ajuda na atualização de um modelo.
anova () - é um teste opcional.
Como criar GLM em R?
Aqui veremos como criar um modelo linear generalizado fácil com dados binários usando a função glm (). E continuando com o conjunto de dados Árvores.
Exemplos
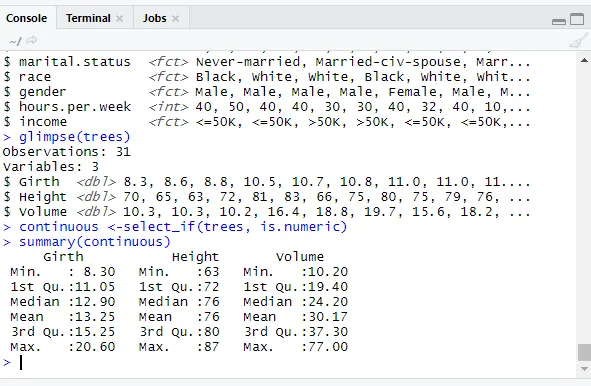
// Importando uma bibliotecalibrary(dplyr)
glimpse(trees)

Para ver os valores categóricos, os fatores são atribuídos.
levels(factor(trees$Girth))
// Verificando variáveis contínuas
library(dplyr)
continuous <-select_if(trees, is.numeric)
summary(continuous)
// Incluindo o conjunto de dados da árvore na pesquisa R Pathattach (árvores)
x<-glm(Volume~Height+Girth)
x
Resultado:
| Chamada: glm (fórmula = Volume ~ Altura + Circunferência)
Coeficientes: (Intercepto) Altura Circunferência -57, 9877 0, 393 4, 7082 Graus de liberdade: 30 total (ou seja, nulo); 28 Residual Desvio nulo: 8106 Desvio residual: 421, 9 AIC: 176, 9 |
summary(x)
| Ligar:
glm (fórmula = Volume ~ Altura + Circunferência) Deviance Residuals: Mín. 1T Mediana 3T Max -6, 4065 -2, 6493 -0, 2876 2.2003 8, 4847 Coeficientes: Estimativa Std. Erro t valor Pr (> | t |) (Interceptação) -57, 9877 8, 6382 -6, 713 2, 75e-07 *** Altura 0, 3393 0, 1302 2, 607 0, 0145 * Circunferência 4, 7082 0, 2643 17, 816 <2e-16 *** - Signif. códigos: 0 '***' 0, 001 '**' 0, 01 '*' 0, 05 '.' 0, 1 '' 1 (Parâmetro de dispersão para a família gaussiana considerada 15.06862) Desvio nulo: 8106, 08 em 30 graus de liberdade Desvio residual: 421, 92 em 28 graus de liberdade AIC: 176.91 Número de iterações de pontuação do Fisher: 2 |
A saída da função de resumo fornece as chamadas, coeficientes e resíduos. A resposta acima mostra que o coeficiente de altura e circunferência não é significativo, pois a probabilidade deles é menor que 0, 5. E há duas variantes de desvio denominadas nulas e residuais. Finalmente, a pontuação de Fisher é um algoritmo que resolve problemas de máxima probabilidade. Com binomial, a resposta é um vetor ou matriz. cbind () é usado para ligar os vetores da coluna em uma matriz. E para obter as informações detalhadas do resumo do ajuste é usado.
Para fazer o teste do Hood, o seguinte código é executado.
step(x, test="LRT")
Start: AIC=176.91
Volume ~ Height + Girth
Df Deviance AIC scaled dev. Pr(>Chi)
421.9 176.91
- Height 1 524.3 181.65 6.735 0.009455 **
- Girth 1 5204.9 252.80 77.889 < 2.2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Call: glm(formula = Volume ~ Height + Girth)
Coefficients:
(Intercept) Height Girth
-57.9877 0.3393 4.7082
Degrees of Freedom: 30 Total (ie Null); 28 Residual
Null Deviance: 8106
Residual Deviance: 421.9 AIC: 176.9
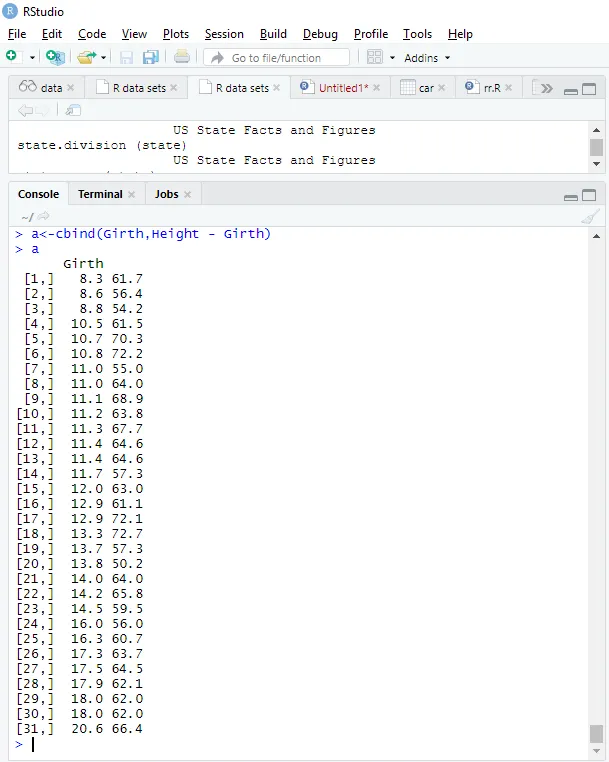
Ajuste do modelo
a<-cbind(Height, Girth - Height)
> a
resumo (árvores)
Girth Height Volume
Min. : 8.30 Min. :63 Min. :10.20
1st Qu.:11.05 1st Qu.:72 1st Qu.:19.40
Median :12.90 Median :76 Median :24.20
Mean :13.25 Mean :76 Mean :30.17
3rd Qu.:15.25 3rd Qu.:80 3rd Qu.:37.30
Max. :20.60 Max. :87 Max. :77.00
Para obter o desvio padrão apropriado
apply(trees, sd)
Girth Height Volume
3.138139 6.371813 16.437846
predict <- predict(logit, data_test, type = 'response')
A seguir, nos referimos à variável de resposta de contagem para modelar um bom ajuste de resposta. Para calcular isso, usaremos o conjunto de dados USAccDeath.
Vamos inserir os seguintes trechos no console do R e ver como a contagem e o quadrado do ano são realizados neles.
data("USAccDeaths")
force(USAccDeaths)
// Analisar o ano de 1973-1978.
disc <- data.frame(count=as.numeric(USAccDeaths), year=seq(0, (length(USAccDeaths)-1), 1)))
yearSqr=disc$year^2
a1 <- glm(count~year+yearSqr, family="poisson", data=disc)
summary(a1)
| Ligar:
glm (fórmula = contagem ~ ano + anoSqr, família = "poisson", dados = disco) Deviance Residuals: Mín. 1T Mediana 3T Max -22, 4344 -6, 4401 -0, 0981 6, 0508 21, 4578 Coeficientes: Estimativa Std. Valor z de erro Pr (> | z |) (Interceptação) 9.187e + 00 3.557e-03 2582, 49 <2e-16 *** ano -7, 207e-03 2.354e-04 -30.62 <2e-16 *** anoSqr 8.841e-05 3.221e-06 27.45 <2e-16 *** - Signif. códigos: 0 '***' 0, 001 '**' 0, 01 '*' 0, 05 '.' 0, 1 '' 1 (Parâmetro de dispersão para a família Poisson considerado 1) Desvio nulo: 7357, 4 em 71 graus de liberdade Desvio residual: 6358, 0 em 69 graus de liberdade AIC: 7149.8 Número de iterações de pontuação do Fisher: 4 |
Para verificar o melhor ajuste do modelo, o seguinte comando pode ser usado para encontrar
os resíduos para o teste. A partir do resultado abaixo, o valor é 0.
1 - pchisq(deviance(a1), df.residual(a1))
Usando a família QuasiPoisson para a maior variação nos dados fornecidos
a2 <- glm(count~year+yearSqr, family="quasipoisson", data=disc)
summary(a2)
| Ligar:
glm (fórmula = contagem ~ ano + anoSqr, família = "quasipoisson", dados = disco) Deviance Residuals: Mín. 1T Mediana 3T Max -22, 4344 -6, 4401 -0, 0981 6, 0508 21, 4578 Coeficientes: Estimativa Std. Erro t valor Pr (> | t |) (Interceptação) 9.187e + 00 3.417e-02 268.822 <2e-16 *** ano -7, 207e-03 2.261e-03 -3.188 0, 00216 ** anoSqr 8.841e-05 3.095e-05 2.857 0, 00565 ** - (Parâmetro de dispersão para a família quasipoisson considerado 92.28857) Desvio nulo: 7357, 4 em 71 graus de liberdade Desvio residual: 6358, 0 em 69 graus de liberdade AIC: NA Número de iterações de pontuação do Fisher: 4 |
Comparando Poisson com o valor AIC binomial difere significativamente. Eles podem ser analisados por precisão e taxa de recuperação. O próximo passo é verificar se a variação dos resíduos é proporcional à média. Em seguida, podemos plotar usando a biblioteca ROCR para melhorar o modelo.
Conclusão
Portanto, focamos no modelo especial chamado modelo linear generalizado, que ajuda a focar e estimar os parâmetros do modelo. É principalmente o potencial para uma variável de resposta contínua. E vimos como o glm se encaixa nos pacotes integrados do R. São as abordagens mais populares para medir dados de contagem e uma ferramenta robusta para técnicas de classificação utilizadas por um cientista de dados. A linguagem R, é claro, ajuda a executar funções matemáticas complicadas
Artigos recomendados
Este é um guia para o GLM em R. Aqui discutimos a função GLM e como criar o GLM em R com exemplos e saída de conjuntos de dados em árvore. Você também pode consultar o seguinte artigo para saber mais -
- R Linguagem de Programação
- Arquitetura de Big Data
- Regressão logística em R
- Trabalhos de análise de big data
- Regressão de Poisson em R | Implementando a regressão de Poisson